CellCelector Software
Benutzerfreundliche und leistungsstarke Software für die automatisierte Detektion und Isolierung von Einzelzellen und Kolonien
Der ALS CellCelector™ ist ein sehr vielseitiges und flexibles System zur automatisierten Analyse und Isolierung von einzelnen Einzelzellen und Kolonien. Dies erfordert eine ebenso flexible und leistungsfähige Software, die den Zugriff auf die zahlreichen Funktionen des Gerätes ermöglicht.
Eine intuitive und einfach zu bedienende grafische Benutzeroberfläche führt den Anwender durch jeden Schritt des Experiments. Unterschiedliche Benutzerzugriffsebenen ermöglichen einen einfachen Zugang zur Software ohne intensive Schulung für neue Anwender und bieten gleichzeitig erweiterte Funktionen, einen offenen Zugang zu allen Parametern, Kalibrierungen und Anpassungsmöglichkeiten für Power-User. Somit wird sowohl eine einfache tägliche Nutzung als auch die Durchführung anspruchsvoller und komplexer Experimente ermöglicht.
Standardarbeitsablauf des CellCelector™ :
Die Benutzeroberfläche der CellCelector™ Software ist an den standardmäßigen Versuchsablauf des Systems angeglichen, indem das Experiment in 5 verschiedene Arbeitsschritte unterteilt ist:
- Scannen von Zellkulturplatten und -schalen zur Aufnahme und Speicherung von Bildern in allen erforderlichen Kanälen (Hellfeld, Phasenkontrast und Fluoreszenz)
- Analyse dieser Bilder und Detektion der Zellen oder Kolonien, die von Interesse sind
- (optional) Überprüfung der Analyseergebnisse durch den Anwender
- Picken der detektierten Einzelzellen und Kolonien
- Dokumentation und Export der Versuchsergebnisse
Jeder Versuchsschritt wird in einer eigenen Benutzeroberfläche angezeigt, die den Zugriff auf genau die richtigen Funktionen und Daten für den jeweiligen Schritt ermöglicht. Alle Optionen und Funktionen, die nicht mit dem aktuellen Versuchsschritt zusammenhängen, sind für den Bediener verborgen, was die Komplexität reduziert.
Scannen
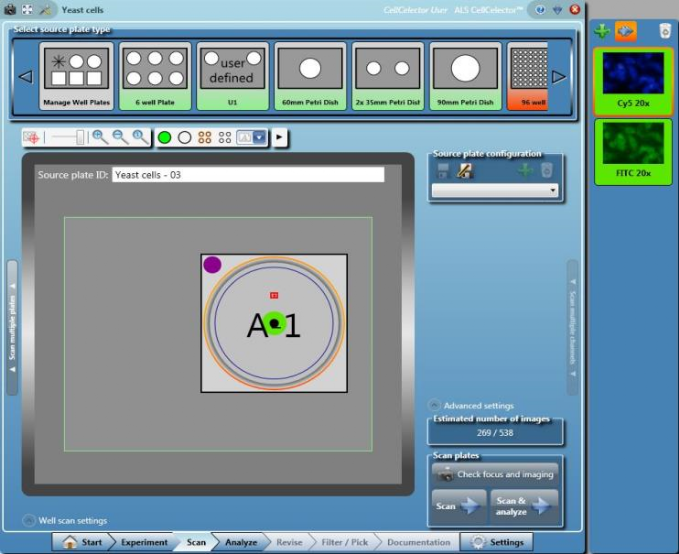
Die Platten und Schalen, die die Probe enthalten, werden automatisch von einer hochauflösenden Kamera auf einem motorisierten Mikroskoptisch gescannt. Die Platten werden aus einer Plattenbibliothek ausgewählt, die die gängigsten kommerziellen Plattenformate enthält und auch die Erstellung von kundenspezifischen Formaten ermöglicht. Zusätzliche Platten, Schalen oder Objektträger können bei Bedarf einfach hinzugefügt werden.
Je nach Anwendung können Scans im Hellfeld, im Phasenkontrast oder in bis zu 6 Fluoreszenzkanälen durchgeführt werden. Alle Belichtungsparameter, von Kameraeinstellungen wie z.B. Belichtungszeit bis hin zu Hardwareeinstellungen wie Filtereinstellungen und Belichtungsparameter, werden in vordefinierten Beleuchtungskonfigurationen gespeichert. Dies ermöglicht das Umschalten zwischen verschiedenen Imaging Setups mit einem einfachen Mausklick. Für das Scannen kann eine beliebige Kombination von vordefinierten Beleuchtungskonfigurationen gewählt werden, um multispektrale Bilder über eine Vielzahl von Kanälen und Beleuchtungsmethoden aufzunehmen.
Alle gespeicherten Bilder des Scans werden von der Software zu einem Übersichtsbild des gescannten Bereiches zusammengefasst:

Analyse und Detektion
Während dieses Schrittes werden alle gespeicherten Bilder analysiert und Zielzellen bzw. Kolonien automatisch auf Grundlage der vom Anwender definierten Parameter erkannt.
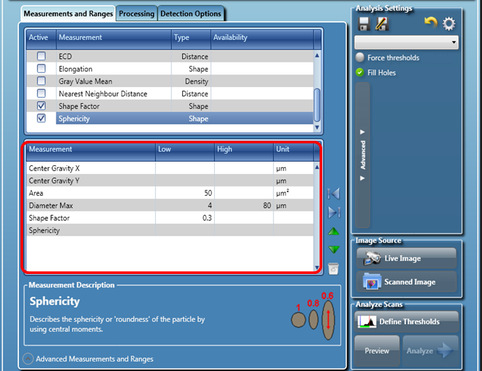
Für jede detektierte Zelle oder Kolonie in jedem aufgenommenen Kanal berechnet die Software ein Panel von morphologischen Messungen wie Fläche, Durchmesser, Form, Sphärizität (Rundheit) etc. Die Verwendung von Filterbereichen für diese Parameter ermöglicht es, die Analyseergebnisse zu filtern, um gezielt nur Zielzellen und Kolonien zu detektieren.
Eine benutzerfreundliche Vorschaufunktion ermöglicht die Überprüfung und Anpassung der Analyseeinstellungen in Echtzeit.
Alle Parameter können als vordefinierte Analyseeinstellungen gespeichert werden und somit eine einfache Routineanalyse von Standardexperimenten ermöglichen.
Review/Überprüfen der Ergebnisse (optional)
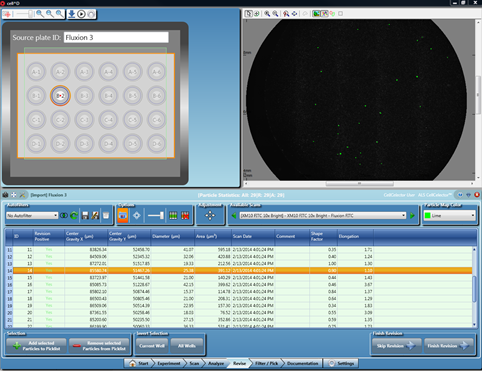
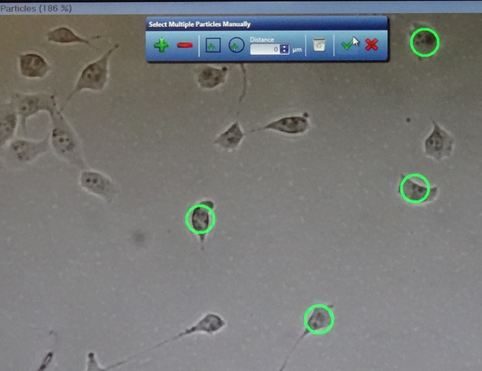
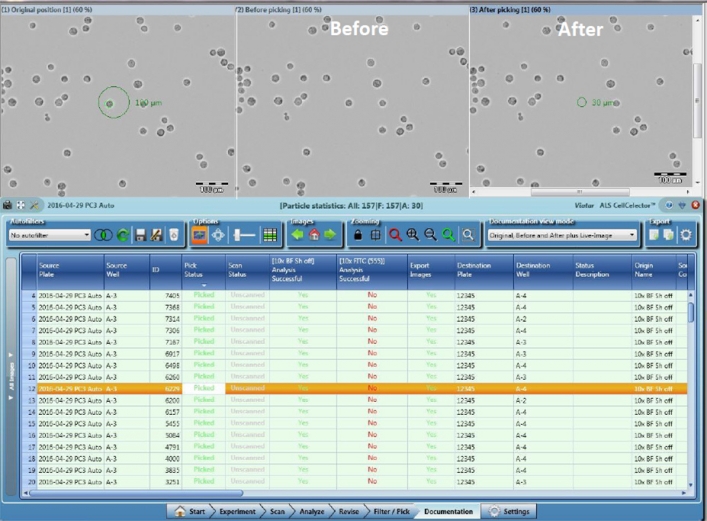
Während der Analyse generiert die Software eine Liste aller detektierten Objekte, die alle gemessenen Daten für jede detektierte Zelle oder Kolonie anzeigt. Bei Bedarf können diese Listen nun vom Anwender überarbeitet werden, indem zusätzliche numerische Filter angewendet oder die Bilder der detektierten Zellen in einer Bildergalerie betrachtet werden. Es ist auch möglich, der Pickliste zusätzliche Elemente hinzuzufügen, indem interessante Objekte direkt am Bildschirm ausgewählt werden.
Picken der Zielobjekte
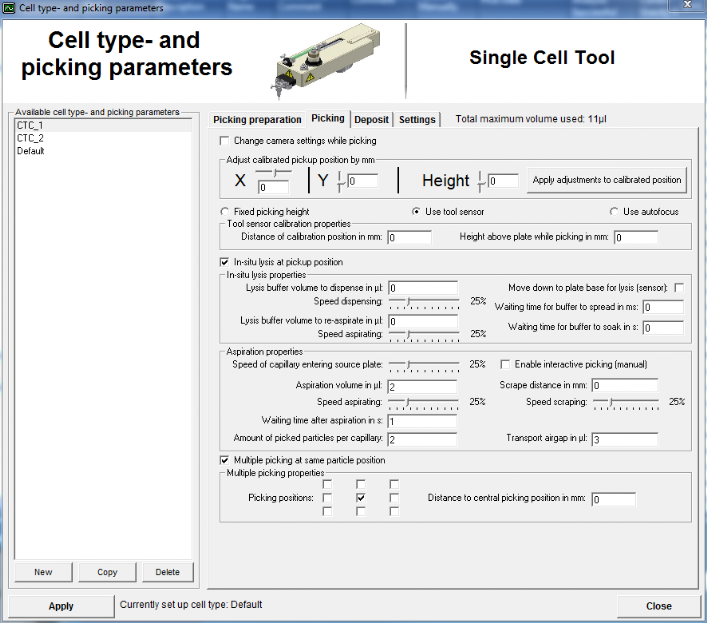
Der ALS CellCelector™ ermöglicht einen sehr schonenden Pickvorgang mit hohen Lebensfähigkeitsraten der entnommenen Zellen oder Kolonien. Daher eignet sich das Gerät hervorragend für die weitere Zellkultivierung, das Klonen von Einzelzellen oder verschiedene molekulare Analysemethoden.
Die Software unterstützt dies, indem sie dem Anwender Zugriff auf eine Vielzahl von Pickparametern (z.B. Aufsaug- und Abgabegeschwindigkeiten, Volumina, Pickhöhe usw.) bietet. Diese Parameter können individuell angepasst und optimiert werden, um ideale Pickergebnisse für Einzelzellen, für Klone aus Suspensions- oder halbfesten Medien als auch für adhärente Kolonien zu erzielen.
Durch die Optimierung dieser Parameter können Objekte nicht nur aus Standard-Zellkulturschalen oder Wellplatten, sondern auch von Membranen, Filtern oder Objektträgern entnommen werden.
Während des Pickvorgangs werden die Zellen von ihrer Ausgangsposition bis zu ihrer Zielplatte und ihrem Zielwell zurückverfolgt. Die Software zeigt während des Pickens ein Live-Bild, wodurch der Anwender den Pickvorgang mikroskopisch verfolgen kann, was ihm eine einfache Optimierung der Pickparameter ermöglicht.
Zur Qualitätskontrolle speichert die Software automatisch Bilder unmittelbar vor und nach jeder Entnahme und ermöglicht so eine spätere Überprüfung der Entnahmeergebnisse (im Hellfeld und/oder einem beliebigen Fluoreszenzkanal). Optional kann die Software die erfolgreiche Entnahme verifizieren, indem sie automatisch den Status jeder Entnahme in der Pickliste aufzeichnet.
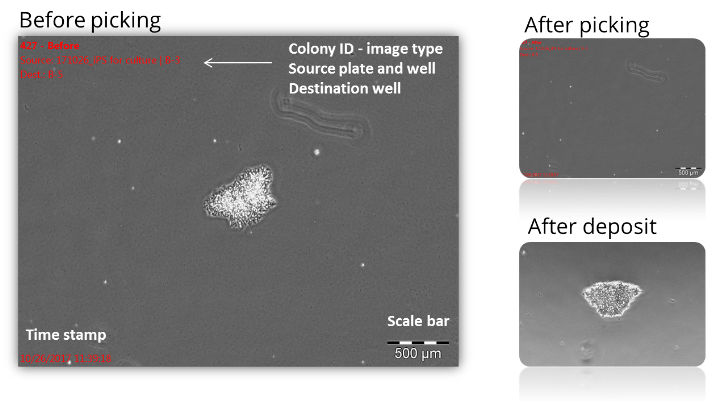
Mikroskopisch hochwertige Bilder werden automatisch vor und nach dem Picken aufgenommen und können einfach exportiert werden.
Die erfolgreiche Ablage der Zielobjekte kann durch das Scannen der Zielplatte visualisiert werden.
Dokumentation
Die vollständigen Daten des Experiments werden auf der Dokumentationsseite der Software GLP-konform aufgelistet und dokumentiert. Alle Daten können zur Dokumentation und Archivierung schnell und einfach exportiert werden.
Jedes detektierte/ausgewählte Objekt kann anhand seiner eindeutigen ID identifiziert und über den gesamten Prozess von der Quelle bis zum Zielort gut verfolgt werden.
Alle bildlichen und numerischen Daten sowie mikroskopisch hochwertige Vor- und Nachheraufnahmen aller entnommenen Zellen und Klone werden in der Datenbank der CellCelector™ Software gespeichert. Ausgangs- und Zielposition jeder Zelle werden in der gleichen Liste erfasst. Auf diese Weise können die Bild- und Pickdaten später, wenn nachgeschaltete Daten (z.B. PCR- oder Kultivierungsdaten) zur Verfügung stehen, einfach wieder aufgerufen werden.
Vorteile und Eigenschaften der CellCelector™ Software
- Perfekt geeignet für die Anforderungen von Forschungs- und Entwicklungsanwendungen durch die Erstellung eigener Protokolle und den offenen Zugang zu allen Parametern
- Einfache Bedienung durch einen grafischen Experimentablauf und die Verwendung von Standardprotokollen für die Routinearbeit
- Hohe Flexibilität durch eine Vielzahl von Parametern für eine qualitativ hochwertige Bilddarstellung, eine ausgefeilte Bildverarbeitung und -analyse sowie für eine sanfte und präzise Isolierung der Zielzellen
- Lückenlose und GLP-konforme Dokumentation des gesamten Prozesses
- Datenverfolgung für jedes Objekt von der Ausgangs- bis zur Zielplatte
- Frei definierbarer Export aller Versuchsdaten zur späteren Dokumentation und Archivierung
- Kontinuierliche Verbesserung der Software durch Anpassung an die Bedürfnisse und Vorschläge unserer Kunden durch unser eigenes Softwareentwicklungsteam
Möchten Sie mehr erfahren?



